种群历史动态分析主要是为了理解种群的进化历史,所利用的数据是重测序数据经过与参考基因组比对过的bam文件进行后续的分析与计算。基本原理是:由于重组的存在,基因组被打断成许多小的片段,利用高深度重测序数据,我们可以找出这些小片段上的变异位点,进而可以推断这些小片段之间的最近共祖时间(the time since the most recent common ancestor,TMRCA)。有的片段之间相似度高,最近共祖时间比较短,有的片段之期差异度比较高,最近共祖时间就比较长。计算基因组各个片段的最近共祖时间,就可以得到最近共祖时间的分布。而最近共祖时间的分布情况包含群体大小随着时间的推移变化信息,根据最近共祖时间分布的比例,可以推算该群体在历史各个时期有效群体大小。我在这里还是跟着组学大讲堂的脚本练习,下面是我自己跑过的流程。
记录跑过的流程
######################################################
#种群动态分析 PSMC
########################################################
cd $workdir #回到工作目录
mkdir 06.PSMC
cd 06.PSMC
#生成psmc需要的染色体一致性序列
## bcftools参数说明
## -Ou : 输出未压缩的bcf文件
## -I : skip indels
## -f reference fasta
## -c 使用原始版本的的calling算法(对应于新的-m)
## -Ov :输出未压缩vcf文件
## -d -D :depth的上下限
bcftools mpileup -Ou -I -f $REF $datadir/CG9203-2.sort.dedup.bam| \
bcftools call -c -Ov| vcfutils.pl vcf2fq -d 10 -D 100| gzip - > CG9203-2.psmc.fq.gz
fq2psmcfa -q20 CG9203-2.psmc.fq.gz > CG9203-2.psmc.fa
##保留染色体上的结果,短片的上的数据去掉
#生成染色体ID列表
cat $FAI |grep "Chr"|cut -f1 >chr.list
#提取染色体数据
get_fa_by_id.pl chr.list CG9203-2.psmc.fa CG9203-2.Chr.psmc.fa
##psmc分析
#-N 迭代次数
psmc -N25 -t15 -r5 -p "4+25*2+4+6" -o CG9203-2.psmc CG9203-2.Chr.psmc.fa
##绘图输出
# -g为该物种繁殖一代的时间,比如人的默认为25年,该处值为-g 25.
# -u 为该物种突变速率
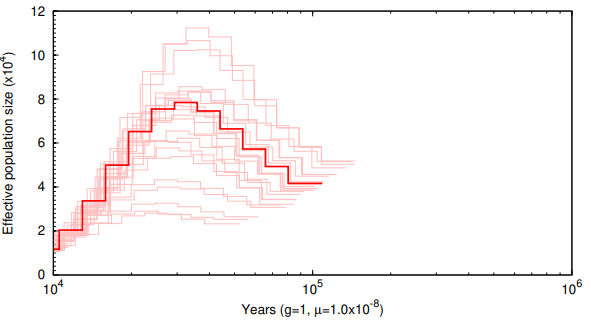
psmc_plot.pl -u 1e-8 -g 1 -p CG9203-2 CG9203-2.psmc
##bootstrapping
##统计序列长度
awk '/^>/{if (l!="") print l; print; l=0; next}{l+=length($0)}END{print l}' CG9203-2.Chr.psmc.fa
splitfa CG9203-2.Chr.psmc.fa 100000 > CG9203-2.split.psmcfa
seq 20 | xargs -i echo psmc -N25 -t15 -r5 -b -p "4+25*2+4+6" -o round-{}.psmc CG9203-2.split.psmcfa >boot.sh
parallel -j 10 <boot.sh # 5个一起并行运行
cat CG9203-2.psmc round-*.psmc > CG9203-2.combined.psmc
#绘图输出
# -g为该物种繁殖一代的时间,比如人的默认为25年,该处值为-g 25.
# -u 为该物种一代的突变速率
psmc_plot.pl -u 1e-8 -g 1 -p CG9203-2.bs CG9203-2.combined.psmc
结果如下: